| Vendredi 29 novembre 2024 | Suivre |
affiner votre recherche
QUALITÉ
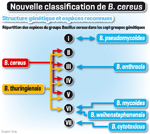
MICROBIOLOGIE Pathogènes Nouvelles avancées
CHANTAL URVOY - RIA - n°717 - novembre 2010 - page 61
Si vous êtes abonné au magazine RIA et déjà inscrit sur RIA.fr
> Renseignez votre identifiant et votre mot de passe.
Si vous êtes abonné au magazine RIA et ne vous êtes pas encore inscrit sur RIA.fr
> Munissez-vous de votre numéro d’abonné qui se trouve sur votre facture et sur le film plastique qui protège votre revue (exemple : A0000005). Puis inscrivez-vous
Si vous n’êtes pas abonné, découvrez gratuitement
> Notre revue papier pendant 2 mois ainsi que tous les contenus du site RIA.fr
Abonnez-vous en ligne
> Vous recevrez chaque mois la revue RIA et profiterez de l’intégralité des contenus du site RIA.fr
Parrainer un ami
> Vous pouvez faire découvrir gratuitement RIA à un ami
Pour ne rien manquer
de la semaine agroalimentaire
recevez gratuitement chaque vendredi
RIA Digest. Infos, emplois, services.
Oui je souhaite m'inscrire afin de recevoir
la newsletter RIA Digest
Je confirme mon mail :
Nos offres d'abonnement à RIA
simples ou couplées.
Découvrir l'offre d'abonnement
 Imprimer
Imprimer